Schnelle manuelle Nukleinsäureisolation mit Spin-Filter-Methode
Die Nukleinsäureisolation mit unseren XXprep-Kits funktioniert nach der Spin-Filter-Methode. Diese Technik ist die Extraktionsmethode der Wahl für geringe Probenmengen und einen regelmäßigen oder auch gelegentlichen Durchsatz von 1-20 Isolationen (low throughput). Mit einer Extraktionsdauer von ca. 20 – 40 min pro Aufreinigung und – je nach Nukleinsäuretyp – einer Ausbeute von bis zu 100 µg hochreiner DNA/RNA ist die „Miniprep“ die Standard-Methode in fast allen molekularbiologischen Laboren (Akademische Forschung, Molekulardiagnostik, Genetic Engineering).
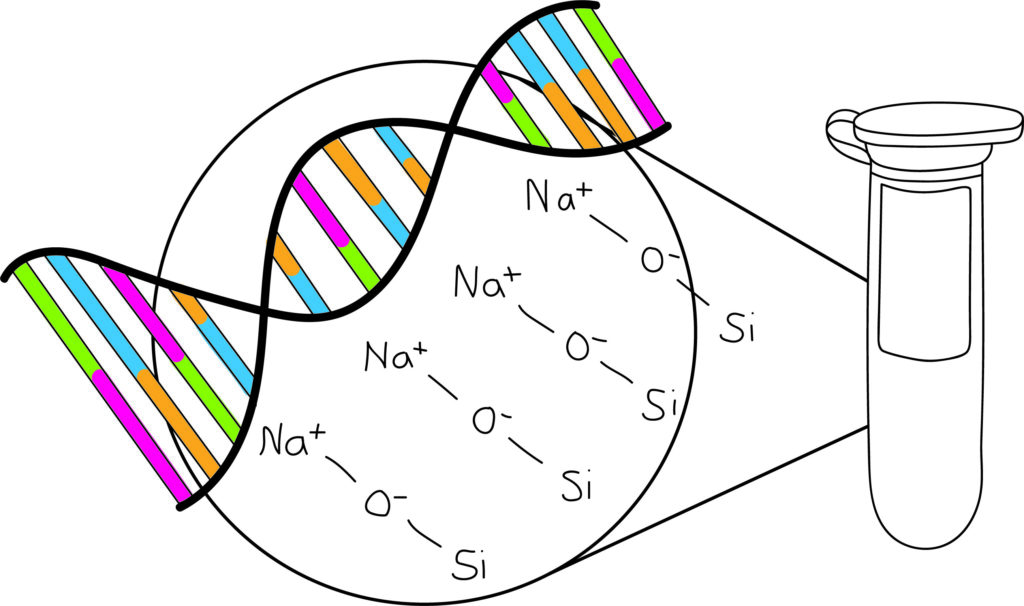
Worauf kommt es bei der Wahl des passenden Kits an?
Für die Anwender*innen spielen Faktoren wie Schnelligkeit der Durchführung, Einfachheit des Protokolls, Vorhandensein aller erforderlichen Gerätschaften (hier lediglich eine Tischzentrifuge), und die Sauberkeit der isolierten Nukleinsäuren eine Rolle. Das Kit muss dabei natürlich zur Probe passen! Es macht einen großen Unterschied, ob ich Plasmid-DNA aus Bakterien oder RNA aus Abwasser isolieren möchte. Die Chemie und das Protokoll müssen optimal aufeinander abgestimmt sein und variieren mit Nukleinsäure- und Probentyp zum Teil erheblich.
Unsere XXprep-Kits für die manuelle Nukleinsäureisolation decken aktuell folgende Nukleinsäuretypen und Ausgangsmaterialien ab:
- genomische DNA aus Bakterien, Gewebe, Nagerschwänzen, eukaryotischen Zellen, Vollblut
- Plasmid-DNA/ Cosmid-DNA aus Bakterien
- Total RNA aus Bakterien, eukaryotischen Zellen und Gewebe
- DNA-Fragmente aus Agarosegelen, PCR-Reaktionen und anderen enzymatischen Ansätzen
Alle XXprep Kits erreichen bei den isolierten Nukleinsäuren eine sehr hohe Reinheit. Typische A260/A280-Werte liegen bei 1.8-1.9 für DNA und 2.0 für RNA.
Wie funktioniert’s?
Die Nukleinsäureisolation mit den XXprep-Kits geschieht immer mittels Spin-Filter-Methode. Das heißt, während es bei der Lyse und Vorbehandlung des Lysats noch große Unterschiede zwischen den Kits gibt, so läuft der Prozess auf der Säule im Großen und Ganzen identisch ab:
- Die Bindung an der Nukleinsäure an die feste Phase erfolgt mit einer Kombination aus chaotropen und nicht-chaotropen Salzen niedriger Ionenstärke. Na+-Ionen vermitteln dabei die Bindung zwischen der negativen Oberfläche der Silica-Matrix (SiO–) und den Phosphatgruppen der Nukleinsäuren.
- Durch das Waschen mit Niedrigsalz-Puffern werden Verunreinigungen von der Säule gespült.
- Die Elution erfolgt unter salzfreien Bedingungen. Die eluierten Nukleinsäuren sind so perfekt für jegliche Art der downstream Applikationen geeignet, z.B. Klonierungen (Restriktionsverdau), PCR, Sequenzierungen, Transfektion und in-vitro Transkription.
XXprep-Kits: Unsere Vorteile
- einfache Durchführung nach Spin-Filter Methode
- Hohe Ausbeute
- Sehr hohe Reinheit
- Reduzierter Kunststoffeinsatz
Übersicht: Die wichtigsten Details zu den einzelnen XXprep Kits
Artikel 4427, Plasmid Mini Kit zur schnellen Isolierung von Plasmid- und Cosmid-DNA aus Bakterienkulturen von bis zu 15 ml.
- reduzierter Kunststoffeinsatz
- neuartige Filter-Membran mit erhöhter Bindekapazität ermöglichen Ausbeuten von bis zu 80 μg hochreiner Plasmid DNA (high-copy und low-copy Plasmide)
- hohe Ausbeuten und hervorragende Reinheit
Die Plasmid-DNA wird durch ein alkalisches Lyseverfahren aus den Bakterienzellen gewonnen und (im Anschluss an die Präzipitation von genomischer DNA und Proteinen) an die Silica-Membran einer Spin-Säule gebunden. Durch verschiedene Waschschritte wird die Plasmid-DNA von Verunreinigungen befreit und anschließend mit 50 – 100 µl Puffer eluiert.
Technische Daten:
Ausgangsmaterial
- Bakterienkultur von 0.5 – 15 ml
Bindekapazität der Säule
- 80 µg DNA
Zeitaufwand
- 15 min
Ausbeute
- 6 – 20 µg (high-copy Plasmid) aus 2 ml Bakterienkultur
- 40 – 80 µg (high-copy Plasmid) aus 15 ml Bakterienkultur
Artikel 2710, Kit für die schnelle und effiziente Isolierung genomischer DNA aus verschiedenen Mengen und Arten an Ausgangsmaterial, wie Gewebeproben, Maus- oder Rattenschwänzen, Bakterien- und eukaryotischen Zellpellets, sowie Vollblut.
- Vielzahl an Ausgangsmaterialien
- reduzierter Kunststoffeinsatz
- Protokolle für 200 μl und 400 μl Vollblut
- hohe Ausbeuten und hervorragende Reinheit
Das Extraktionsverfahren kombiniert die Lyse unterschiedlichster Ausgangsmaterialien mit der anschließenden Reinigung der DNA über eine Silica-Säule.
Achtung: Mit dem Standard-Protokoll wird neben DNA auch RNA isoliert. Anwender*innen, die RNA-freie DNA benötigen, können optional einen RNase-Verdau vor dem Aufbringen der Probe auf die Säule durchführen.
Technische Daten:
Ausgangsmaterial
- Gewebeproben bis zu 20 mg
- Vollblut (frisch oder gefroren; stabilisiert mit EDTA oder Citrat) bis zu 400 µl
- Nagerschwanzstücke 0,2 – 0,8 cm
- Zellkulturen bis zu 5 x 106 Zellen
- Bakterienkulturen bis zu 1010 Zellen
Bindekapazität der Säule
- 100 µg DNA
Zeitaufwand
- nach Lyse ca. 10 min
Ausbeute
- Abhängig vom Probenmaterial; kann mehr als 100 µg DNA pro Säule betragen
Reinheit
- A260/A289: 1.8 – 2.0
- A260/A230: 1.8 – 2.3
Artikel 5482, Kit für die schnelle Aufreinigung von PCR Produkten im einfachen Zwei-Schritt-Verfahren und/oder für die Extraktion und Rückgewinnung von DNA-Fragmenten aus TAE oder TBE-Agarosegelen.
- Kombi-Kit
- PCR -Aufreinigung im Zwei-Schritt-Verfahren (ohne überflüssiges Waschen)
- reduzierter Kunststoffeinsatz
- flexible Elutionsvolumina (10 – 50 μl)
- geeignet für Fragmentlängen von 100 bp bis 30 kb
- hohe Ausbeuten und hervorragende Reinheit
Eigentlich genauso wie bei der genomischen DNA, nur dass hier ein spezieller Bindepuffer zum Einsatz kommt, der für kleinere DNA-Stücke optimiert wurde. Außerdem ist die Oberflächenfunktionalisierung der Silica-Matrix eine andere.
Technische Daten:
Ausgangsmaterial
- PCR- und Sequenzierreaktionsmischungen bis zu 50 µl
- TAE oder TBE-Agarosegele bis zu 300 mg
- Fragmentlängen 100 bp – 30 kb
Bindekapazität der Säule
- 20 µg DNA
Zeitaufwand
- PCR- und Sequenzieraufreinigung ca. 3 min (2-Schritt-Verfahren)
- Gelextraktion ca. 20 min
Rückgewinnungsraten
(Abhängig von Fragmentlänge)
- PCR-Aufreinigung 60 – 95 %
- Sequenzierungs-Aufreinigung min. 75 %
- Gelextraktion 60 – 90 %
Artikel 6780, Kit für die schnelle und effiziente Isolierung von zellulärer Gesamt-RNA aus eukaryotischen Zellen, Bakterien und Gewebeproben.
- Vielzahl an Ausgangsmaterialien
- Selektive Entfernung genomischer DNA ohne DNase-Verdau
- kein giftiges und übelriechendes ß-Mercaptoethanol erforderlich
- reduzierter Kunststoffeinsatz
- hohe Ausbeuten und hervorragende Reinheit
Ein speziell optimiertes Lysepuffersystem garantiert die Isolierung intakter RNA bei dauerhafter Deaktivierung endogener und exogener RNasen. Mittels einer Vorsäule wird die genomische DNA entfernt, was den DNase I-Verdau überflüssig macht. Die RNA wird an eine zweite Silica-Säule gebunden, gewaschen und schließlich in 30 – 80 μl mit RNase-freiem Wasser eluiert.
Achtung: Das Kit ist nicht für virale RNA geeignet! Zur Lyse von Viren braucht es ein anderes Lysepuffersystem.
Technische Daten:
Ausgangsmaterial
- Gewebeproben (Biopsien) bis zu 20 mg
- Zellkulturen bis zu 5 x 106 Zellen
- Bakterienkulturen bis zu 109 Zellen
Bindekapazität der Säule
- 100 µg RNA
Zeitaufwand
- 15 – 40 min
Ausbeute
- Abhängig vom Probenmaterial; kann bis zu 100 µg RNA pro Säule betragen
Reinheit
- A260/A289: 1.8 – 2.1

